Estimation du dN/dS et
détection de l'adaptation
0. Programme
Nous allons utiliser codeml, un des programmes du paquet PAML de Ziheng
Yang, le prorgamme le plus utilisé pour estimer le dN/dS. Codeml
a été installé sur vos machines mais est
téléchargeable ici pour
une utilisation ultérieure (vous trouverez également des
instructions pour son installation).
Codeml permet d'estimer le dN/dS via
des analyses par sites, par branches ou les 2. Il permet
également de faire de tester la significativité des
résultats via des tests de rapport de vraisemblance.
Nous allons traiter 2 examples qui couvrent les principales
fonctionalités de codeml et qui montrent l'intêret de ce
programme pour la détection de l'adaptation au niveau
moléculaire.
1. Adaptation du virus HIV-1 au système immunitaire humain
Le système de défense contre HIV-1 chez l'homme repose
sur la production par l'organisme d'anticorps et de lymphocytes T
cytotoxiques (LTC), qui est elle-même régulée par
les lymphocytes T helpers (TH). Un élément de cette
défense (les LTH) sont la cible de HIV-1 et leur
élimination progressive entraine l'apparition de maladies
opportunistes chez les patients atteints du SIDA. Le succès de
HIV-1 réside dans sa capacité d'évader à la
reconnaissance par le système immunitaire. Le
séquençage de virus à différentes phases de
l'infection a montré une augmentation des mutations
non-synonymes au niveau des épitopes des LTC qui suggère
de la sélection positive sur ces sites. Des observations
indirectes suggèrent que le même phénomène a
lieu également pour les épitopes reconnus par les
anticorps. La maximisation de la variation des antigènes de
surface de HIV-1 semble être la clef de sa capacité
à échapper au système immunintaire. Afin de tester
cette idée et de mieux comprendre la variation
antigénique de HIV-1, nous allons étudier la
sélection positive dans le génome de HIV-1.
a) HIV-1 contient différents gènes: gag, pol, vif, vpr, env. Les
fichiers ci-dessous contiennent les séquences de 26 souches de
HIV-1 (et incluent notamment les types principaux A, B et C):
GAG.phy
POL.phy
VIF.phy
VPR.phy
ENV.phy
b) Vous allez lancer codeml dans un premier temps sur le gène env en utilisant l'arbre
phylogénetique de ces 26 souches:
HIV_1.tree
c) Lancer une analyse par sites avec les modèles suivants: M0,
M1, M2a, M3, M7, M8 en modifiant le fichier suivant:
codeml.ctl
d) Comparer les modèles suivants: M0 et M3, M1 et M2a, M7 et M8,
qu'en déduisez vous?
e) Lancer les analyses sur les autres gènes.
e) Quelles analyses supplémentaires proposeriez vous?
Pour plus d'information:
Yang W, Bielawski JP, Yang Z (2003) J Mol Evol 57: 212-21.
2. Divergence fonctionnelle des copies de la chalcone synthase, une
enzyme impliquée dans la pigmentation des fleurs
Les chalcones synthases (CHS) sont des enzymes clefs de la
biosynthèse des flavonoides, qui sont important pour la
pigmentation des fleurs et donc la pollinisation. Les gènes
codant pour les CHS font partie de familles multigéniques dont
le nombre varie entre plantes et dont les copies peuvent varier
fonctionnellement. Chez les ipomées, 5 gènes CHS (A-E)
ont été décrits. Par ailleurs, les ipomées
sont caractérisées par une grande diversité de
couleurs de leurs fleurs. Par exemple, le volubilis (Ipomoea purpurea) possède
des fleurs blanche, rose, bleue ou bleue foncé. Ces
diffèrences sont sous-tenues par des diffèrences au
niveau de la synthèse des flavonoides et probablement des CHS
qui catalyzent la première étape de la voie de
synthèse des flavonoides. Plusieurs études indiquent une
divergence fonctionnelle importante au sein des CHS, par exemple les
stilbènes synthases (STS) semblent avoir émerger des CHS,
ainsi que les acridones synthases (ACS), les bibenzyles synthases
(BBS), les 2-pyrones synthases (2PS) et les phlorisovalerophenones
synthases (PVPS). Pour tester si les CHS ont divergé
fonctionnellement et acquis de nouvelles fonctions, nous allons les
analyser avec codeml.
a) Les fichiers suivant contiennent les séquences
alignées de 45 CHS de plantes dont un échantillon de CHS
A, B, C, D et E de diffèrentes espèces d'ipomées,
et leur arbre phylogénétique:
CHS.phy
CHS.tree
b) Lancer codeml sur ces données en modifiant le fichier suivant:
codeml.ctl
c) Réaliser une analyse par branches en étiquetant les
branches indiquées dans l'arbre ci-dessous et avec 1 dN/dS,
2 dN/dS (branche a et le reste), 7 dN/dS
(toutes les branches/lignées indiquées plus le reste).
Comparer les différents modèles.
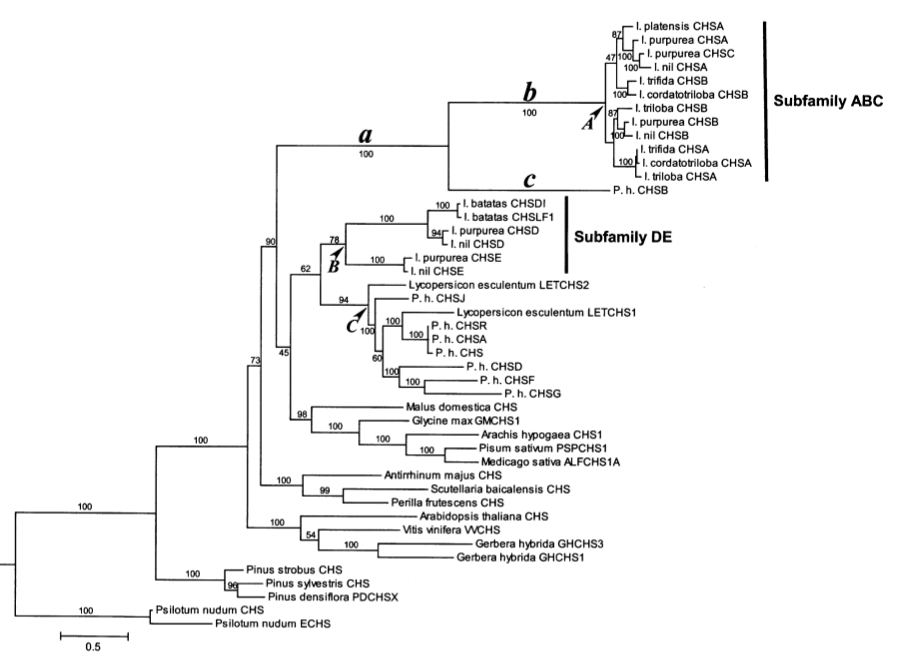
d) Réaliser une analyse par branches et par sites sur les
branches/lignées montrant des dN/dS
élevés dans l'analyse par branches, qu'en déduisez
vous? Tester la significativité des résultats.
c) Quelles analyses supplémentaires proposeriez vous?
Pour plus d'information:
Yang J, Gu H, Yang Z (2004) J Mol Evol 58:54-63.
