Sommaire de la page
Thèmes de recherche
Le nom du laboratoire met en évidence les deux thèmes fédérateurs : Biométrie et Biologie évolutive. Ainsi le laboratoire s’organise autour d’un point de vue méthodologique qui affirme l’importance de la modélisation tant mathématique qu’informatique et d’une perspective évolutive qui organise les recherches indépendamment du niveau d’organisation biologique. L’originalité du laboratoire est donc qu’il s’affirme être à la fois un laboratoire de biométrie et un laboratoire de biologie. Le maintien de l’équilibre entre ces deux aspects est un enjeu fort de la politique scientifique du laboratoire, c’est en effet dans la synergie entre des problématiques biologiques propres et des développements méthodologiques que naît la plus grande part des résultats scientifiques du laboratoire. Les conséquences de cette politique se sont exprimées en termes de moyens, d’équipements et de postes, elles seront détaillées dans la description de la structure du laboratoire.
Il n’est pas question ici de décliner l’ensemble des recherches qui s’articulent autour de ces deux axes. Cette description sera trouvée dans les textes de chacun des départements. On peut simplement souligner ici que, comme tous les biologistes, nous nous intéressons à des systèmes complexes mais que l’approche méthodologique choisie permet de mettre cette complexité au cœur de la recherche. Ainsi les phénomènes d’interaction entre organismes au sein de relations de parasitisme ou symbiose, entre niveau d’organisation dans la relation entre génétique des populations et fonctionnement génomique, entre organisme et environnement tant dans le cadre de systèmes écologiques que de systèmes liés à la santé ont été au centre de nos recherches.
Cette période contractuelle a revêtu une importance particulière dans le cadre des relations sciences/santé. C’est en effet ce contrat qui a intégré officiellement dans la structure d’UMR l’équipe de J. Estève, actuellement dirigée par P. Roy, et donc le thème « modélisation en épidémiologie » sur le versant santé. Cette association nous a paru être un succès, et les perspectives que nous proposons conduisent à un renforcement du lien entre ces deux aspects du Vivant au sein du laboratoire.
Un deuxième aspect très important dans l’évolution des thèmes scientifiques du laboratoire a été la création de l’équipe Baobab autour de M..F. Sagot. M.F. Sagot a été nommé en 2001 CR INRIA au sein du laboratoire dans le cadre du projet INRIA Helix auquel participait l’équipe « Bionformatique et Génomique évolutive » animée par M. Gouy. Le passage DR de M.F. Sagot en 2003 a conduit à la création d’une équipe au sein d’Helix regroupant les chercheurs les plus « informaticiens » dans le domaine du génome. Cette équipe renforce les relations du laboratoire avec la recherche en informatique et constitue donc un aspect important de notre politique scientifique.
Structure du laboratoire
Le précédent contrat (1998-2002) avait vu le nombre de chercheurs et d’enseignants chercheurs du laboratoire passé de 36 à 49. À la fin de ce contrat, ce nombre sera de 59 (compte non tenu des demandes d’intégrations pour le futur contrat). L’augmentation régulière de la taille du laboratoire se poursuit, quant au personnel de recherche, la quasi-stagnation des personnels d’aide à la recherche se poursuit également. En prenant en compte l’ensemble des personnels ITA et IATOS, toutes fonctions confondues, leur nombre est passé de 14 en 1998 à 16 en 2002 pour atteindre actuellement 16 en 2005. Le nombre de personnel technique par personnel de recherche a donc pris au fil des derniers contrats les valeurs de 0,39 - 0,33 - 0,27. Si la politique des tutelles reste de dire que la priorité est de développer le soutien technique à la recherche, les chiffres ont du mal à plaider pour l’efficacité de cette politique. Dix ans de demandes, de revendications, d’argumentations ont usé notre espoir de réussir à rétablir une situation normale sur ce point qui constitue certainement le plus grand échec de la direction de laboratoire.
L’organisation technique du laboratoire s’appuie de plus en plus sur des plateaux techniques internes et des plates-formes externes. Deux plates-formes jouent un rôle important pour le laboratoire :
- Le DTAMB
- (Développement Technologique et Analyse Moléculaire de la Biodiversité, Dir. J.J.Madjar) offre des possibilités de spottage et lecture de puces à ADN, de séquençage et analyse de fragments. Le DTAMB est une plate-forme de l’IFR 41, et plate-forme Génopole pour sa partie puces à ADN
- Le PRABI
- (Pôle Rhône-Alpes de BioInformatique, Dir. C. Gautier) est une plate-forme opérationnelle RIO et une plate-forme Génopole.
À ces plates-formes doit être ajouté le Centre de Calculs de l’IN2P3 qui nous permet la réalisation de l’ensemble des calculs intensifs.
Le laboratoire a cependant besoin de moyens propres d’acquisition et de traitement de données. Pour ces moyens propres, nous avons essayé de mettre en œuvre une politique de mutualisation avec la création de plateaux techniques internes. Le plus ancien concerne l’informatique avec des serveurs communs, et un service informatique (voir organigramme) qui gère ces serveurs et le réseau du laboratoire. De nouveaux plateaux sont apparus dans le cadre de la biologie moléculaire soit autour d’équipements (microscope à fluorescence) soit autour de techniques communes (service de PCR en cours de mise en place). D’autres sont en cours de gestation et émergeront après la fin des travaux concernant les locaux du PRABI.
Une des conclusions importante du précédent rapport d’évaluation du laboratoire concernait le manque criant de locaux. Nous étions en 2000 et un projet CPER venait d’être accepté pour la construction d’un nouvel étage principalement consacré à la plateforme PRABI. La réactivité remarquable de nos instances de gestion fait que, au moment de l’écriture de ces lignes, le laboratoire a toujours le même espace, que les problèmes de locaux que nous considérions comme à la limite du soutenable ont pu cependant augmenter mais …. le bruit plein d’espoir du marteau piqueur résonne, enfin.
Au cours de ce contrat nous avons expérimenté l’organisation en départements, cette expérimentation a été positive sur le principe, mais sans doute pas totalement optimale quant au découpage choisi. D. Mouchiroud, en accord avec le conseil de l’UMR, a choisi de poursuivre ce type d’organisation (voir la partie perspectives) mais dans un découpage différent. Nous avons choisi d’organiser l’ensemble du bilan sur la base de ce nouveau découpage, ceci peut paraître illogique, le bilan se référant au passé. Cela nous a cependant paru plus facile pour évaluer le potentiel futur du découpage proposé. Ainsi le bilan sera présenté en 3 départements « verticaux » à savoir : BioMaths-Santé (BMS), Écologie Évolutive (EE) et Génétique et Génomique Évolutives (GGE). Deux axes transversaux assurent la cohésion globale du laboratoire : l’axe « interactions durables » (ID) est un choix scientifique qui sera discuté dans les perspectives, son bilan sera très court puisqu’il s’inscrit plus dans le futur que le passé, l’axe « méthodologie » (METH) reconnaît en fait une activité ancienne et souligne l’importance que cette activité joue dans le cadre de la communication entre les champs d’application. Le bilan de METH sera donc aussi détaillé que ceux des départements verticaux.
Bibliométrie
Le paragraphe bibliométrie est indispensable dans un rapport d’activité scientifique ; il est sensé présenter de manière objective la production d’un laboratoire. Bien entendu, comme tout résumé de données, il n’est pas objectif mais objectivé pour montrer le laboratoire sous son meilleur jour. Le jeu entre évalués et évaluateurs étant clair nous ne nous poserons pas de problèmes déontologiques ici, par contre les valeurs citées sont exactes aux difficultés près de recollement informatique des travaux d’un laboratoire. En théorie les règles utilisées ont été :
- Seules sont comptabilisées (sauf pour l’analyse sur les records) les publications réalisées dans le laboratoire. Cette règle est la plus difficile à suivre car le statut dans et hors laboratoire des personnels change avec le temps ; malgré cela s’il y a des erreurs, elles sont rares et non biaisées.
- Le département BMS est, actuellement, uniquement analysé au travers des facteurs d’impact, la dispersion sur plusieurs sites et la dépendance de nombreuses tutelles n’a pas rendu possible une interrogation fiable des sites de bibliométrie.
- Enfin ce travail a pu être réalisé ici grâce à l’accès aux moyens dont tout directeur de laboratoire devrait disposer, ce qui est loin d’être le cas général. Il est scandaleux que d’une part les politiques nous assènent des indices défavorables à la recherche française et d’autre part ne nous donnent pas les outils d’analyse dont tout laboratoire européen dispose pour analyser l’impact de sa production.
Analyse par journal
Les trois tableaux ci-dessous présentent les 15 revues ayant les plus forts impacts facteurs pour chacun des départements. Ces tableaux montrent bien l’originalité des thèmes de chacun des départements avec très peu de recouvrement dans les journaux cibles. L'ensemble des publications classées par département et journaux est présentée ici
| journal | nombres d'articles | Impact Facteur |
|---|---|---|
| Journal of Animal Ecology | 9 | 2,843 |
| Ecological Applications | 1 | 2,852 |
| Journal of Evolutionary Biology | 2 | 3,01 |
| Oecologia | 5 | 3,128 |
| Journal of Applied Ecology | 1 | 3,205 |
| Banach Center Publications, Polish Academy of Science | 1 | 3,436 |
| Ecology | 8 | 3,701 |
| Applied and Environmental Microbiology | 1 | 3,820 |
| Molecular Ecology | 2 | 3,870 |
| American Naturalist | 3 | 4,059 |
| Ecology Letters | 2 | 4,211 |
| Emerging Infectious Diseases | 1 | 5,340 |
| Neuroscience and Biobehavioral Reviews | 1 | 5,482 |
| Trends in Ecology and Evolution | 2 | 12,449 |
| journal | nombres d'articles | Impact Facteur |
|---|---|---|
| BMC BIOINFORMATICS | 2 | 5,423 |
| MOLECULAR BIOLOGY AND EVOLUTION | 9 | 6,050 |
| JOURNAL OF BIOLOGICAL CHEMISTRY | 1 | 6,482 |
| ONCOGENE | 1 | 6,495 |
| NUCLEIC ACIDS RESEARCH | 9 | 6,575 |
| BIOINFORMATICS | 7 | 6,701 |
| CURRENT OPINION IN MICROBIOLOGY | 2 | 8,182 |
| HUMAN MOLECULAR GENETICS | 1 | 8,597 |
| GENOME RESEARCH | 4 | 9,635 |
| PROCEEDINGS OF THE NATIONAL ACADEMY OF SCIENCES OF THE UNITED STATES | 2 | 10,452 |
| CURRENT OPINION IN GENETICS & DEVELOPMENT | 1 | 11,58 |
| TRENDS IN GENETICS | 1 | 12,016 |
| PLOS BIOLOGY | 1 | 13,868 |
| SCIENCE | 1 | 29,781 |
| NATURE | 1 | 30,979 |
| journal | nombres d'articles | Impact Facteur |
|---|---|---|
| HUMAN REPRODUCTION | 1 | 3.125 |
| INTERNATIONAL JOURNAL OF SYSTEMATIC AND EVOLUTIONARY MICROBIOLOGY | 2 | 3.187 |
| JOURNAL OF CLINICAL MICROBIOLOGY | 1 | 3.489 |
| AMERICAN HEART JOURNAL | 1 | 3.681 |
| EUROPEAN JOURNAL OF CANCER | 2 | 3.694 |
| APPLIED AND ENVIRONMENTAL MICROBIOLOGY | 2 | 3.820 |
| ANTIMICROBIAL AGENTS AND CHEMOTHERAPY | 1 | 4.246 |
| AMERICAN JOURNAL OF SURGICAL PATHOLOGY | 3 | 4.535 |
| JOURNAL OF CEREBRAL BLOOD FLOW AND METABOLISM | 3 | 5.370 |
| CLINICAL INFECTIOUS DISEASES | 2 | 5.594 |
| EMERGING INFECTIOUS DISEASES | 1 | 5.643 |
| AIDS | 1 | 5.893 |
| ARCHIVES INTERN MED | 2 | 7.508 |
| BRAIN | 2 | 7.967 |
| CANCER RES | 1 | 8.649 |
Histogramme pour les trois départements des facteurs d’impact
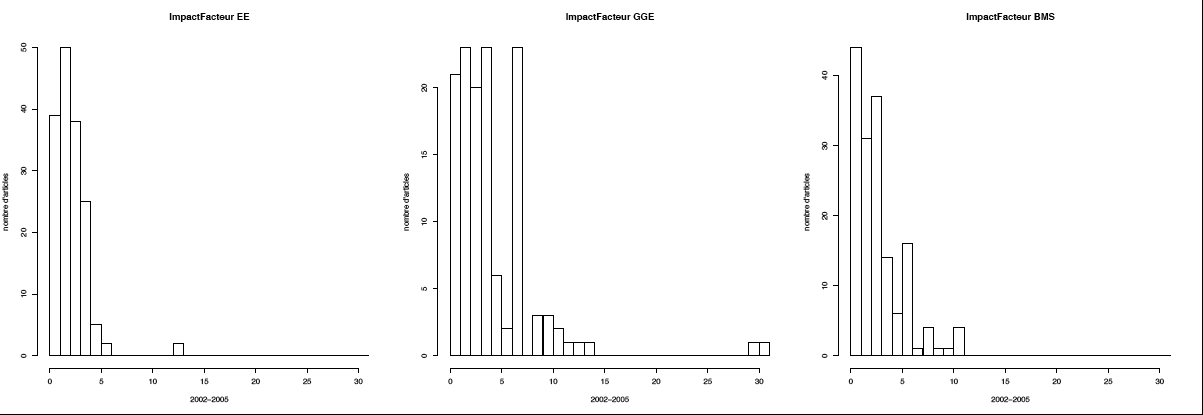
Analyse du nombre de citations
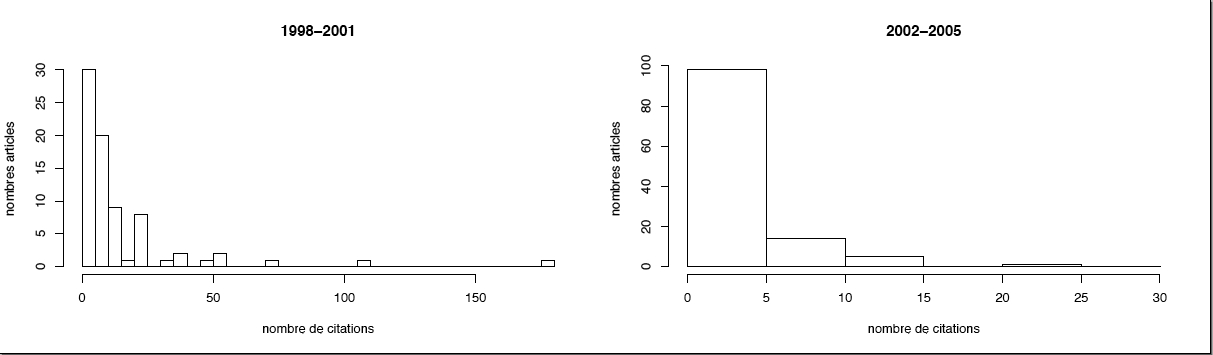
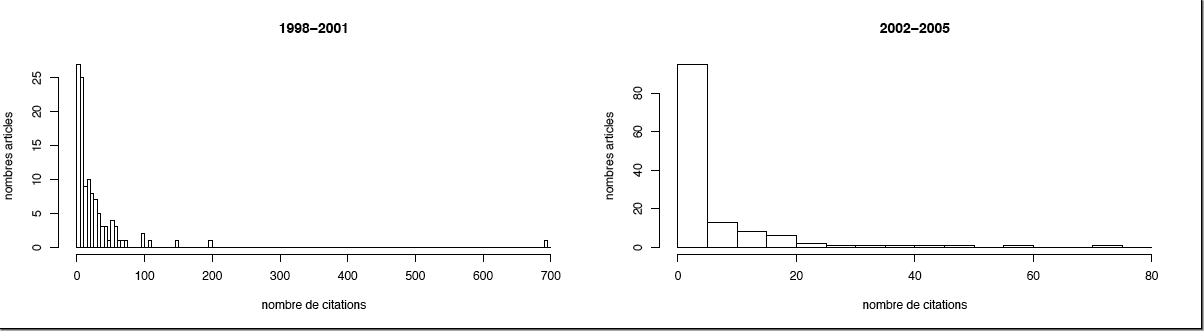
Un des critères soulignés par le ministère dans les objectifs et en tant qu’indicateur de la recherche est « l’indice de citation à deux ans des opérateurs du programme ». Nous nous sommes inspirés de cette remarque en l’étendant quelque peu. Pour chacun des départements dans leurs nouveaux contours (mais en ne prenant bien entendu en compte que les membres présents dans le laboratoire au moment de la publication) nous avons cherché le nombre de citations de chacune des publications. Ceci a été réalisé sur deux périodes correspondant au contrat en cours et au contrat précédent. Bien entendu les chiffres pour le contrat en cours ne peuvent qu’être faible, les articles étant jeunes et le temps de citation très court. Le résultat est résumé dans les histogrammes ci-dessous :
Ces mêmes résultats sont résumés dans les deux tableaux suivants correspondant chacun à une des périodes d’étude.
| Dpt | 1er Quartile | Médiane | Moyenne | 3e Quartile | Maximum |
|---|---|---|---|---|---|
| EE | 3,0 | 7,0 | 15,1 | 15,0 | 179,0 |
| GGE | 6,0 | 14,0 | 29,4 | 29,0 | 694 |
| Dpt | 1er Quartile | Médiane | Moyenne | 3e Quartile | Maximum |
|---|---|---|---|---|---|
| EE | 0 | 1 | 2,7 | 3 | 25 |
| GGE | 0 | 2 | 6,3 | 6,5 | 72 |
Analyse des records
La dernière analyse se fonde sur les personnels actuellement membres du laboratoire quel que soit leur statut actuel et passé. Seuls les articles ayant été cités plus de 50 fois ont été pris en compte. Bien évidemment cette statistique avantage, a priori les plus âgés (ce qui me semble assez juste !), elle donne cependant une idée globale de l’impact des personnels du laboratoire dans le domaine scientifique.
département Ecologie Evolutive :
département Génomique et Génétique Evolutives
Ces mêmes résultats sont résumés dans les deux tableaux suivants correspondant chacun à une des périodes d’étude.
| Dpt | 1er Quartile | Médiane | Moyenne | 3e Quartile | Maximum |
| EE | 3,0 | 7,0 | 15,1 | 15,0 | 179,0 |
| GGE | 6,0 | 14,0 | 29,4 | 29,0 | 694 |
Relations Internationales
Les relations internationales s’organisent autour de contrats et de publications communes. Les contrats impliquant des partenaires étrangers ont été regroupés dans « Contrats avec partenariat à l’étranger » p. 236 du rapport écrit. Les différents pays concernés sont lagement réparti dans le monde: Asie (Viet Nam, Japon), Europe (contrat européen, Russie, Irlande, Espagne, Norvège, Suéde), Amériques (Mexique, Argentine, Brésil), Afrique (Algérie, Afrique du Sud). Ils montrent deux orientations principales : la première vers l’Europe et la deuxième vers l’Amérique du Sud. Dans le cas de l’Europe, trois contrats européens et deux Pics organisent ces relations. Par rapport aux contrats, il faut ajouter le fait que l’Institut Suisse de bio-informatique est associé au projet Hélix de l'INRIA auquel les équipes de M. Gouy et MF Sagot appartiennent. Il en est de même avec l'université de São Paulo au Brésil. D'une manière plus générale les relations qu'entretient le labnoratoire avec l'Amérique centrale et l'Amérique du Sud sont fortes. Au relations avec le Brésil de l'équipe Baobab s'ajoute en effet les relations institutionnelles et scientifiques fortes des équipes de C. Bernstein et F. Menu avec le Mexique et l'Argentine. Dans le domaine de l'écologie des vertébrés de nombreuses relations existent avec les pays nordiques (Norvège et Suède en particulier).
Enseignement
La biométrie et la biologie évolutive apparaissent complémentaires aussi bien dans l’activité de recherche que dans l’activité de formation. Ainsi l’équipe pédagogique du laboratoire a été amenée à développer dans ces deux champs disciplinaires des enseignements pour l’ensemble des cycles et impliquant différentes structures présentes sur Lyon (Université, ENS, INSA).
Un des moteurs de notre démarche est le besoin de formation de biologistes ayant une bonne connaissance en mathématiques et en informatique, besoin qui ne s’exprime pas uniquement aux niveaux de la recherche mais aussi au niveau de l’ingéniorat ainsi qu’à des niveaux de sortie du système éducatif inférieur (licence et master).
Offres de formation de l’UMR 5558
L’offre de formation de l’équipe pédagogique regroupe 45 unités d’enseignement scientifiques (16 en licence et 29 en master) qui recouvrent à la fois des champs méthodologiques (bioinformatique, biomathématique, biostatistique) et biologiques (évolution moléculaire, génomique, biologie évolutive, génétique et dynamique des populations). Nous sommes responsables de l’ensemble des enseignements de Mathématiques et d’Informatique pour la biologie. En santé, les enseignants du département BioMaths-Santé sont responsables de la totalité des enseignements de biostatistiques et de microbiologie au niveau des premières années de la formation médicale.
Filière « BIM »
Dans le cadre d’une cohabilitation avec l’ENS Lyon, nous participons depuis plusieurs années à la formation des étudiants de la filière « Biologie Moléculaire et Cellulaire » (ancien magistère) au travers des enseignements de biostatistique, de bioinformatique, d’évolution moléculaire et de génomique. Nous partageons avec un enseignant de l’ENSL, la responsabilité de la licence (http://www.ens-lyon.fr/DSVT/BMC/bmc.html ).
Filière « BMC »
Dans le cadre d’une cohabilitation avec l’ENS Lyon, nous participons depuis plusieurs années à la formation des étudiants de la filière « Biologie Moléculaire et Cellulaire » (ancien magistère) au travers des enseignements de biostatistique, de bioinformatique, d’évolution moléculaire et de génomique. Nous partageons avec un enseignant de l’ENSL, la responsabilité de la licence (voir le site ). Lors de la mise en place du LMD, nous avons proposé en première année du master « BMC » un module d’enseignement européen concernant la génomique comparative « Comparative Genomics » ouvert à la fois aux étudiants lyonnais mais également aux étudiants européens dans le cadre des échanges « ERASMUS ». Le stage pratique de bioinformatique associé à ce module peut être réalisé dans l’une des équipes de recherche associées au GDRE « Comparative Genomic » reconnu par le CNRS en 2005 qui regroupe 21 laboratoires, 29 équipes et 14 instituts et dont le responsable est C. Biémont, DR CNRS.Filière « MIV »
La mise en place du LMD à l’université Lyon 1 (L1 en sept. 2003, le reste du cursus en sept. 2004) nous a donné l’opportunité de créer une nouvelle filière inter disciplinaire, mathématiques, informatique et biologie : MIV ou Mathématiques et Informatique du Vivant. Cette filière s’appuie sur une mention de licence « MIV » (resp. L. Gueguen) et sur un master « aMIV » (Resp. S. Charles et C. Gautier) (voir le site).
Cette filière qui recrute les étudiants à partir de la deuxième année de licence (L2) a pour objectif de donner une formation avec une double compétence « biométrique et biologique » et ceci pour tous les champs de la biologie grâce à des modules optionnels choisis dans les différents parcours de Biologie de l’Université.
Le master « aMIV » ou approches Mathématique et Informatique du Vivant » est ouvert à ces étudiants ayant cette double compétence. Ce master comprend une première année commune ; trois spécialités « recherche » et une spécialité « professionnalisante » sont proposées en deuxième année.
| Recherche | MBM | Méthodes en Bioinformatique Moléculaire | G. Déléage, PU D. Mouchiroud, PU |
| MERC | Modélisation en Épidémiologie et Recherche Clinique | P. Roy, PU-PH | |
| AMIT | Approche quantitative et Modélisation formelle pour l’Innovation Thérapeutique | F. Gueyffier, PU-PH | |
| Professionel | oMIV | Outils Mathématiques et Informatiques pour le Vivant | A-B. Dufour, MCU |
Les différentes spécialités recherche du master aMIV s’appuient sur les compétences méthodologiques et les thématiques de recherche décrites dans les deux départements « Biomaths-Santé » et « Génétique et Génomique Évolutives ».
Il est à noter que certains membres des deux équipes demandant leur intégration dans l’UMR (P. Vanhems et F. Gueyffier participent aux enseignements dispensés dans les deux spécialités MERC et AMIT. Par ailleurs, F. Gueyffier est responsable de la spécialité AMIT.
Master EEME, spécialité EEB
Notre laboratoire est également responsable d’une seconde spécialité de master M2 « Écologie, Évolution et Biométrie » (voir le site) (resp. D. Pontier) qui est dans la continuité du DEA « Analyse et Modélisation des Systèmes Biologiques » mis en place en 1986. L’objectif est de former des étudiants à la recherche dans le cadre de l’écologie, la biologie évolutive et la biométrie en s’appuyant sur l’acquisition de bases méthodologiques fortes et la maîtrise des concepts des théories actuelles.
La spécialité « Écologie, Évolution et Biométrie » tout comme le master aMIV émargent à la même École Doctorale « Évolution Écosystèmes Microbiologie Modélisation » (voir le site) dont le directeur, JP. Flandrois est membre du laboratoire.
Autres formations
Par ailleurs, le laboratoire a créé en 2000 une filière de Master à Ho-Chi-Mhin (Vietnam) en Biométrie (resp. C. Gautier) impliquant chaque année, cinq enseignants du laboratoire. De même MF. Sagot vient de créer au Portugal (ouverture 2005) un master de bioinformatique : « PhD Program in Computational Biology » instituto Gulbenkian de Cienca, Lisbonne.
Afin de donner une lisibilité internationale à notre formation en Bioinformatique, deux initiatives seront entreprises (1) la création d’un Master International de Lyon 1, (2) un master « Erasmus Mundus » de Bioinformatique ouvert sur un recrutement européen et des pays tiers. Ces créations se justifient d’une part en raison des compétences fortes en bioinformatique dans le laboratoire ainsi que dans d’autres établissements lyonnais (IBCP, INSA) et dans la région Rhône-Alpes (INRIA Grenoble) ; d’autre part pour palier la désaffection de nos étudiants pour les cursus orientés vers la recherche.
En raison de l’implication forte du laboratoire dans l’activité de formation, le service informatique (S. Delmotte) a mis en place une base de données « RepServ » permettant la gestion automatisée des services de tous les enseignants-chercheurs, moniteurs, ATER et vacataires. Cette base de données constitue actuellement une référence au sein de l’UFR de Biologie.
2- Technologies de l’information et de la Communication pour l’Enseignement
Le deuxième point central dans la démarche de l’équipe pédagogique est que l’apprentissage des mathématiques en biologie ne soit pas déconnectée de la biologie. Dans ce sens, nous avons mis en place un site WEB dédié à l’enseignement des mathématiques pour les Sciences de la Vie au travers des nouvelles technologies.
Ce site, MathsSV, qui constitue un support à l’enseignement magistral propose un cours intégral d’analyse, d’algèbre et de statistiques ainsi que des exercices corrigés, des annales d’examen et des QCM. L’agenda est une page Web donnant aux étudiants chaque semaine des informations concernant les chapitres du cours à étudier, les exercices ou problèmes à résoudre ainsi que les résultats des contrôles ou des sondages qui ont été effectués. Un forum de discussion permet aux étudiants de dialoguer avec leur enseignant. Parallèlement un site WEB dédié à l’enseignement de la génétique des populations a été développé avec les mêmes caractéristiques.
Un enseignement de biostatistique dédié aux étudiants de niveau L3 et master a été développé sous forme de fiche de cours et travaux dirigés entièrement sous R (voir site). Enfin un atelier de simulation de phénomènes biologiques dynamiques a été implémenté en langage Mathematica (Dynatica).
L’ensemble de ces outils a pour objectif un apprentissage des biomathématiques au travers des nouvelles technologies. Ainsi la création du site MathSV nous a permis de réduire la part des cours magistraux et de mettre en place des travaux tutorés sur machine permettant une aide personnalisée pour les étudiants (petit effectif) avec une connexion directe au cours.
Nous souhaitons dans l’avenir étendre cet aspect de notre formation à d’autres disciplines enseignées par l’équipe pédagogique et ainsi établir des liens entre les enseignements plus méthodologiques (biostatistique, biomathématiques et bioinformatique) et ceux plus biologiques (génétique et biologie des populations, évolution moléculaire, écologie évolutive), complémentarité qui sous-tend la recherche dans notre laboratoire. Ceci se fera dans le cadre de la mise en place d’une plateforme d’enseignement « Biométrie et Biologie Évolutive ».
Il faut insister sur le fait que la mise en place du système LMD a amputé l’activité de recherche des enseignants-chercheurs d’au moins deux années. Il n'est donc pas envisageable que notre équipe pédagogique participe à une quelconque refonte de l'enseignement dans le prochain contrat quadriénal
Responsabilités collectives des membres du laboratoire
Journaux scientifiques :
Editeur : J of Biological System (P. Auger)
- Editeur associé :
- Mol. Biol. Evol (M. Gouy)
- section "Review"de Heredity (C. Biemont) ;
- BMC Ecology (D. Pontier)
- Wildlife Biology (A. Loison)
- IEEE/ACM Transactions in Computational Biology and Bioinformatics (M.F. Sagot)
- BMC Bioinformatics (M.F. Sagot)
- BMC Algorithms in Molecular Biology (M.F. Sagot)
- Computational Biology and Chemistry (M.F. Sagot)
- Lectures Notes in BioInformatics (LNBI) (M.F. Sagot)
- Research in Microbiology (M.F. Sagot)
De plus une dizaine de membres du laboratoire font partie du comité éditorial de journaux internationaux.
Responsabilités locales et régionales :- Présidence de l’Université Claude Bernard – Lyon 1 (D. Debouzie 2001-2006)
- Administrateur des Hospices civils de Lyon (G. Carret)
- Direction de l’École Doctorale E2M2 (J.P. Flandrois)
- Membre du CS de l'Université Lyon 1 (J. Lobry)
- Directeur du DEA AMSB puis Master EEB (D. Pontier)
- Directeur du master aMIV (S. Charles)
- Directeur adjoint de l’UFR de Biologie (M. Boulétreau puis C. Gautier depuis 2003)
- Chef du Service de Biostatistique des HCL (R. Écochard)
- Membre du Conseil Scientifique de l’INVS (R. Écochard)
- Animateur des Plateformes de Bioinformatique (C. Gautier depuis 2004)
- Directeur du PRABI (C. Gautier depuis 2004)
Responsabilités nationales :
- EPST :
- Président de la Commission 30 au Comité national du CNRS (M. Boulétreau, 2000-2004)
- Président de la Commission 29 au Comité national du CNRS (C. Gautier, 2004- 2008)
- Membres de la Commission 30 au Comité national du CNRS (M. Boulétreau, N. Gautier 2000-2004)
- Membres de la Commission 29 au Comité national du CNRS (C. Gautier, F. Fleury, 2004-2008)
- Membres de la Commission 44 au Comité national du CNRS (MF. Sagot)
- Responsable du programme inter EPST « Bioinformatique » (C. Gautier 2003)
- Membre du CS du département BIA de l’INRA (C. Gautier)
- Membre du CS de l’ACI « IMPBIO » (L. Duret)
- Chargé de mission « bioinformatique » du département Science de la Vie du CNRS (C. Gautier jusqu’à 2003)
- EPIC et Agences :
- Président de la commission AFSSA "Risque et Listeria" (J.P. Flandrois 2000-2004)
- Président du Comité d'expert spécialisés "Microbiologie" de l'AFSSA (J.P. Flandrois)
- Ministère de la Recherche et de la Technologie :
- Membre du CNU (M. Gouy)
- Autres :
- Membre du comité de pilotage de la mission parlementaire Cohen-Le Déaut (J.P. Flandrois)
- Membre correspondant de l'Académie des Sciences (P. Auger)
- Membre du CS de l'IFB (M. Gouy)
- Membre du COPE (Comité Opérationnel pour l’Éthique du CNRS) (C. Biémont)
- Directeur du GDR CNRS « Évolution des Éléments transposables : du génome aux populations ») (C. Biémont ).
- Directeur du GDR 2153 « Évolution des relations parasitaires chez les insectes » (F. Vavre)
- Directeur du GDRE « Comparative Genomics » (C. Biémont 2005)
- Membre du Conseil scientifique du groupe FERTILI (Padou, Italie) (R. Écochard)
Hygiène et Sécurité
La sécurité des personnels et de l'environnement a été identifiée comme un objectif de l’UMR au même titre que les objectifs scientifiques ou de formation. Ceci a été exprimé par le Conseil de l’UMR et a été explicité par une Déclaration de la Direction en 1996. Dès lors une « politique sécurité » a été mise en place.
Le Document d’Évaluation de la Sécurité a montré que l’unité est concernée par 20 catégories de risques sur les 22 répertoriés, ceci justifie la mise en place d’une politique efficace de sécurité, mais met l’accent sur les difficultés induites par la polyvalence des équipes. L’UMR a fondé sa politique de sécurité sur l’information, l’encadrement et la mise progressive aux normes de ses équipements.
1) Information
Chaque année lors de la réunion de rentrée des personnels, une sensibilisation à la culture sécuritaire a été faite, elle renvoie à trois documents :
- le manuel de survie du nouvel entrant
- le manuel de sécurité
- le site internet «